Home > News > Une nouvelle microscopie pour observer en 3D les mécanismes cellulaires à l’échelle nanométrique en profondeur
Une nouvelle microscopie pour observer en 3D les mécanismes cellulaires à l’échelle nanométrique en profondeur
by - 3 July 2019
L’observation des systèmes biologiques par microscopie de fluorescence connait une véritable révolution avec l’avènement des techniques de super-résolution optique permettant de dépasser la limite de diffraction, travaux récompensés par le prix Nobel de Chimie en 2014 et le prix Breakthrough en 2018. Le microscope se mue ainsi en nanoscope offrant des échelles d’observation jusque-là inégalées. Cependant différentes limitations restreignaient encore la portée des observations comme une résolution en profondeur relativement limitée et restreinte ainsi que la persistance de différents biais de mesures comme des dérives axiales ou des problèmes de chromatismes limitant les performances de ces microscopes.
Des physiciens de l’Institut des Sciences Moléculaire d’Orsay (ISMO, CNRS/Université Paris Saclay), de l’Institut Langevin (ESPCI/CNRS) et du Centre de Photonique Biomédical (Université Paris Saclay) viennent de montrer que la position axiale des molécules fluorescentes pouvait être obtenue de façon absolue avec une précision nanométrique. Pour cela, ils ont utilisé une propriété particulière de l’émission de fluorescence à proximité de la lamelle de verre sur laquelle les cellules sont déposées. Cette émission dite supercritique est en plus insensible aux dérives axiales ainsi qu’au problème de chromatisme. En associant cette technique à d’autres méthodes de localisation axiales, ils ont pu étendre les performances de localisation au-delà du premier micron, tout en conservant une mesure absolue et sans biais. En collaboration avec des collègues chimistes (ISCN, CNRS/ Université Paris Saclay) et biologistes (INP, CNRS/Université Aix Marseille), cette nouvelle méthode, appelée DAISY, a permis d’observer en 3D différentes protéines du cytosquelette cellulaire, l’insertion d’une sonde fluorescente introduite par « click chemistry » dans des bactéries ou la position de différentes protéines dans des neurones.
Ces résultats viennent d’être publiés dans Nature Communications. Ils sont le fruit d’une collaboration étroite et fortement interdisciplinaire entre des laboratoires du CNRS relevant de l’INP, l’INC, l’INSIS et l’INB. Ils ont reçu le soutien du Fond AXA pour la recherche, du Labex WIFI, du DIM NanoK et du réseau européen LaserLab. Ces travaux font l’objet d’un transfert technologique auprès de la société Abbelight, jeune start-up en biotechnologies issue de l’ISMO et de l’Institut Langevin. De nombreuses recherches en biologie vont ainsi pouvoir bénéficier très rapidement de cette nouvelle technique qui offre des avantages uniques en termes d’imagerie cellulaire 3D.
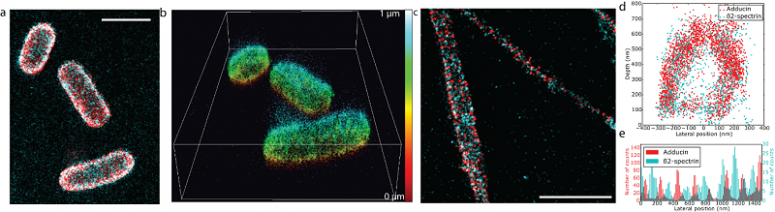
- Figure : a) et b) représentent une image DAISY en 2D et 3D de bactéries E.Coli marquées par click chemistry, c) représente l’organisation de l’adducine et la B2 spectrine dans des neurones de rat, d) et e) sont des coupes axiale et latérale de © qui confirme l’organisation tubulaire et l’alternance latérale des 2 protéines avec une périodicité de 190 nm.
Combining 3D single molecule localization strategies for reproducible bioimaging
C. Cabriel, N. Bourg, P. Jouchet, G. Dupuis, C. Leterrier, A. Baron, M.-A. Badet-Denisot, B. Vauzeilles, E. Fort and Sandrine Lévêque-Fort
Nature Communications 10, 1980 (2019)
Contact :
Sandrine Lévêque-Fort, ISMO, sandrine.leveque-fort@u-psud.fr
Emmanuel Fort, Institut Langevin, emmanuel.fort@espci.fr


