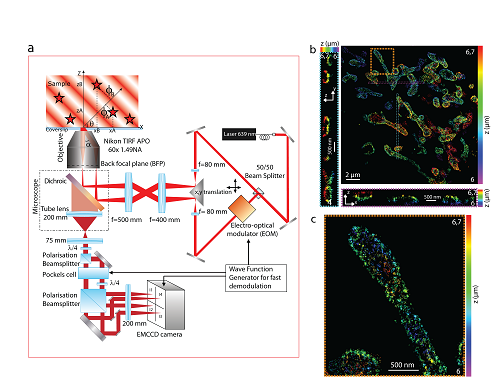
Révéler la position tridimensionnelle de molécules avec des précisions nanométriques uniformes est aujourd’hui possible quelle que soit la profondeur à l’intérieur de l’échantillon biologique. Ces résultats de recherche originaux ont été obtenus par des équipes de l’Institut des Sciences Moléculaires d’Orsay (dans le cadre de la thèse de Pierre Jouchet), de l’Institut Langevin et de l’unité de recherche INSERM 1193, en proposant un nouveau concept dans le processus de localisation des molécules uniques.
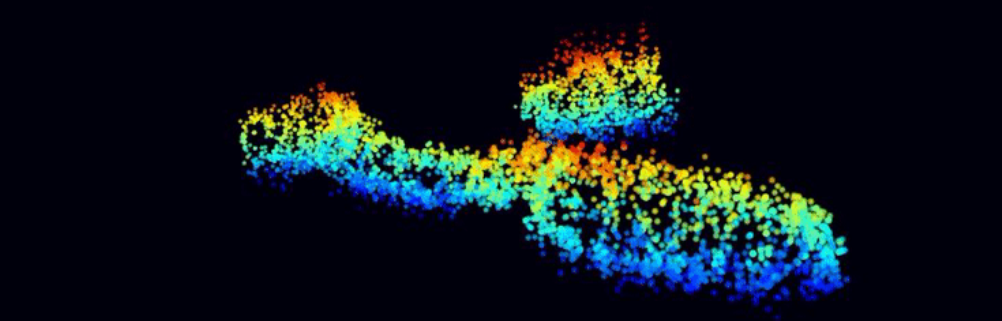
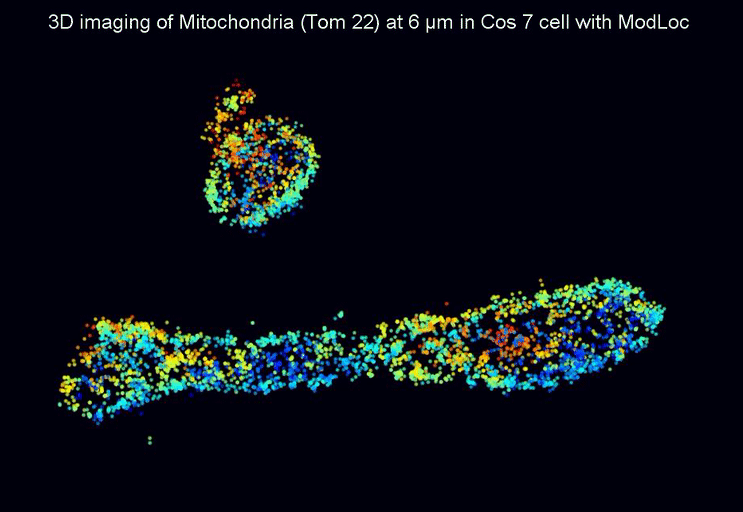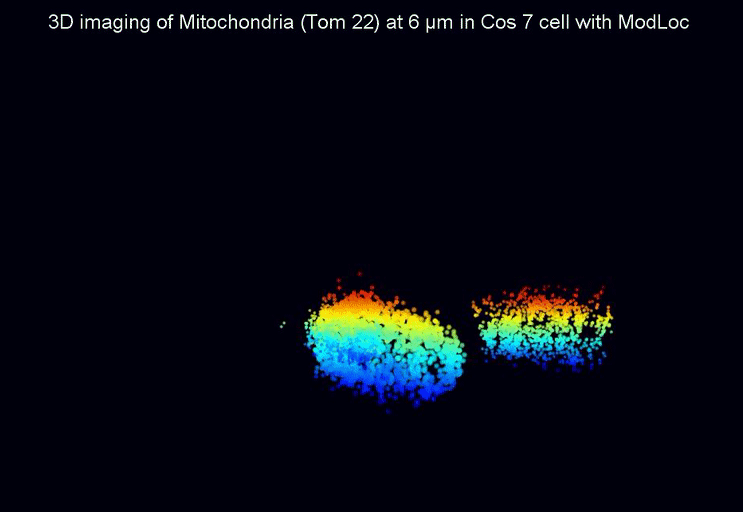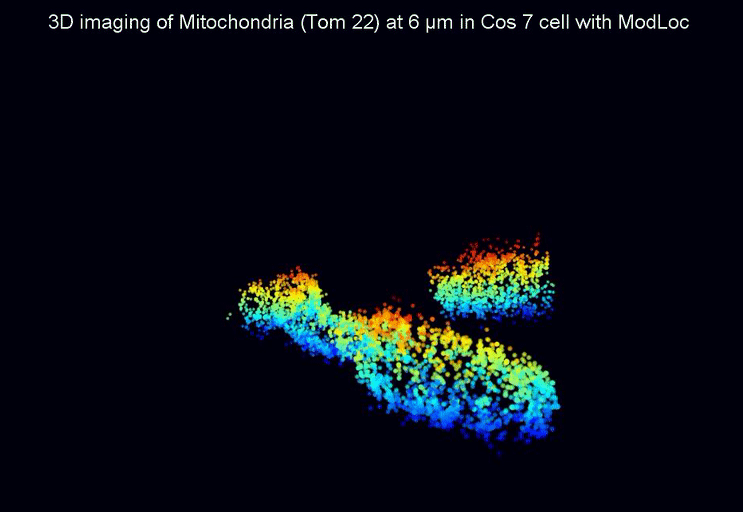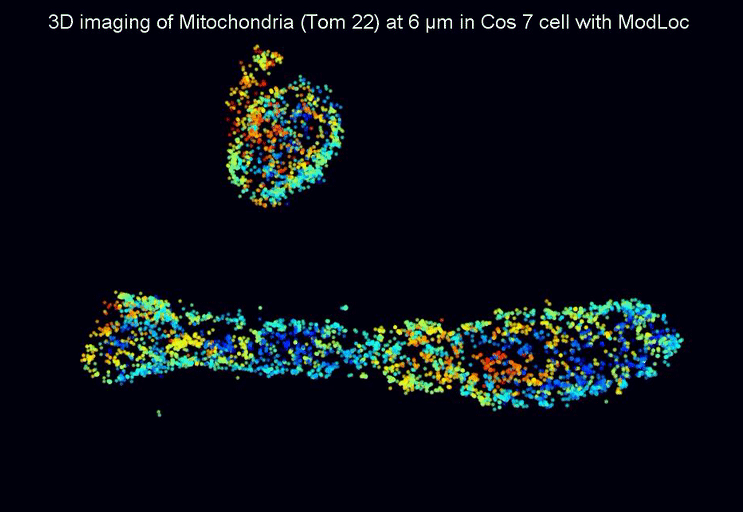
En substituant l’illumination uniforme de l’échantillon classiquement utilisée, par une illumination structurée variant rapidement au cours de temps, l’émission de fluorescence des molécules est alors modulée temporellement, et révèle intrinsèquement la position de chacune des molécules. Cette technique appelée ModLoc pour « Modulated Localization », permet d’atteindre des précisions axiales proches de la taille des biomolécules (6.8 nm). Les développements en microscopie optique dite de super-résolution (récompensée par le prix Nobel de chimie en 2014) visent à pouvoir reproduire de façon fidèle l’organisation en 3D des protéines localisées à plusieurs dizaines de micromètres dans des échantillons complexes. En introduisant ce paramètre d’observation temporelle, les chercheurs ont pu montrer que la technique ModLoc permettait d’atteindre une résolution uniforme jusqu’à plusieurs dizaines de microns, alors que pour l’ensemble des techniques jusque-là proposées, la résolution variait avec la profondeur d’observation, de plus le champ d’observation proposé reste compatible avec les applications biologiques visées. ModLoc peut s’implémenter sur tout microscope optique et ouvre ainsi de nouvelles perspectives pour la biologie fondamentale ou le diagnostic médical, où le besoin de comprendre la localisation en 3D de molécules est un enjeu majeur de la recherche d’aujourd’hui. Protégé par un brevet CNRS, ce nouveau concept permettant d’extraire un paramètre de l’échantillon avec une précision constante à l’échelle nanométrique, peut se décliner pour l’observation d’autres informations que la position des molécules, et a d’ores et déjà fait l’objet d’une étude de valorisation.
Publication sur le site de Nature :
Jouchet, P., Cabriel, C., Bourg, N. et al. Nanometric axial localization of single fluorescent molecules with modulated excitation. Nat. Photonics (2021). https://doi.org/10.1038/s41566-020-00749-9